Teilbereich 1: Altern von Stammzellen
Die Forschungsgruppen innerhalb des Teilbereichs 1 untersuchen die Ursachen und Folgen der Stammzellalterung. Die Forschungsarbeit erstreckt sich von Modellorganismen über genetische Mausmodelle bis hin zu Mausmodellen, die mit menschlichen Stammzellen angereichert sind.
Mit der Schließung von zwei Gruppen in 2016 ist die Stammzellforschung an Wirbellosen-Modellen im Teilbereich 1 reduziert. Das Institut geht davon aus, dass mit der Rekrutierung neuer Gruppen diese Lücke geschlossen warden kann.
Die Forschung ist durch vier Schwerpunkte definiert:
- Zellintrinsische Mechanismen, die die Funktion alternder Stamm- und Vorläuferzellen einschränken,
- Alternsbedingte Veränderungen der Stammzellnischen und des systemischen Umfelds,
- Mechanismen der klonalen Selektion und epigenetische Drifts bei der Stammzellalterung und
- Mikrobiota- und stoffwechselbedingte Beeinträchtigungen der Stammzellfunktion während des Alterns (im Zusammenhang mit dem neuen Schwerpunkt Mikrobiota und Altern, der ím Teilbereich 2 aufgebaut wird).
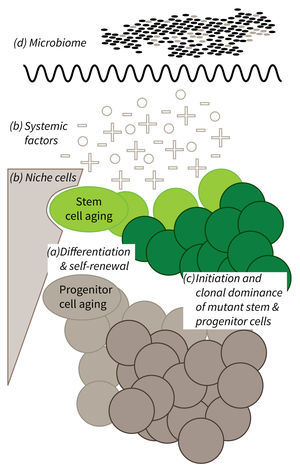
Forschungsfokus von Teilbereich 1.
a) Derzeit ist noch nicht vollständig bekannt, welche Mechanismen die Zellfunktionen im Alter beeinträchtigen. b) Der relative Einfluss von Nischenzellen und systemisch wirkenden Faktoren auf die Stammzellalterung müssen für verschiedene Gewebe noch erforscht werden. c) Die klonale Ausbreitung mutanter Zellen wird mit der Entstehung von Krankheiten im Alter in Verbindung gebracht. Mechanistisch ist der Prozess jedoch noch kaum verstanden. Die Veränderungen in der Farbintensität stehen für die klonale Dominanz von ursprünglichen Stamm- (grün) und Vorläuferzellen (grau). d) Es gibt zunehmend Hinweise darauf, dass alternsbedingte Veränderungen im Mikrobiom die Stammzellfunktion beeinflussen und vice versa.
Publikationen
(seit 2016)
2025
- The protein deacetylase HDAC10 controls DNA replication in malignant lymphoid cells.
Mieland AO, Petrosino G, Dejung M, Chen JX, Fulzele A, Mahmoudi F, Tu JW, Mustafa AHM, Zeyn Y, Hieber C, Bros M, Schnöder TM, Heidel FH, Najafi S, Oehme I, Hofmann I, Schutkowski M, Hilscher S, Kosan C, Butter F, Bhatia S, Sippl W, Krämer OH
Leukemia 2025, 39(7), 1756-68 - Embryonic macrophages orchestrate niche cell homeostasis for the establishment of the definitive hematopoietic stem cell pool.
Perçin** G, Riege K, Fröbel J, Metz J, Culemann S, Lesche M, Reinhardt S, Höfer T, Hoffmann S, Waskow** C
Nat Commun 2025, 16(1), 4428 ** co-corresponding authors - Malignant JAK-signaling: at the interface of inflammation and malignant transformation.
Perner F, Pahl HL, Zeiser R, Heidel FH
Leukemia 2025, 39(5), 1011-30 - Mesenchymal Stromal Cells regulate human Hematopoietic Stem Cell survival, engraftment and regeneration via PGE2/cAMP signaling pathway
Sai Naga Anurag Muddineni S, Katz Even C, Zipin-Roitman A, Rasoulouniriana D, Singha Roy D, Aborgies R, Beider K, Raz Y, Solomon N, Hershkovitz G, Shulman Y, Chen R, Weizman E, Waskow C, Nagler A, Milyavsky M
bioRxiv 2025, https://doi.org/10.1101/2023.11. - Anti-CD19 CAR-T cell therapy for acquired hemophilia A.
Schultze-Florey CR, Thol FR, Aleksandrova K, Stoyanov K, Gutierrez Jauregui R, Arseniev L, Leise J, Klöß S, Heidel** FH, Tiede** A
Leukemia 2025, 39(4), 980-2 ** co-corresponding authors - Enhancer Hijacking Discovery in Acute Myeloid Leukemia by Pyjacker Identifies MNX1 Activation via Deletion 7q.
Sollier E, Riedel A, Toprak UH, Wierzbinska JA, Weichenhan D, Schmid JP, Hakobyan M, Touzart A, Jahn E, Vick B, Brown-Burke F, Kelly K, Kelekçi S, Pejkovska A, Goyal A, Bähr M, Breuer K, Chen MJM, Llamazares-Prada M, Hartmann M, Schönung M, Correia N, Trumpp A, Abdullah Y, Klingmüller U, Mughal SS, Brors B, Westermann F, Ulrich E, Autry RJ, Schlesner M, Vosberg S, Herold T, Greif PA, Pfeifer D, Lübbert M, Fischer T, Heidel FH, Gebhard C, Walter W, Haferlach T, Eisfeld AK, Mrózek K, Nicolet D, Bullinger L, Smeenk L, Erpelinck-Verschueren C, Mulet-Lazaro R, Delwel R, Ernst A, Scherer M, Lutsik P, Jeremias I, Döhner K, Döhner H, Lipka DB, Plass C
Blood Cancer Discov 2025, 6(4), 343-63
2024
- Imetelstat-mediated alterations in fatty acid metabolism to induce ferroptosis as a therapeutic strategy for acute myeloid leukemia.
Bruedigam C, Porter AH, Song A, Vroeg In de Wei G, Stoll T, Straube J, Cooper L, Cheng G, Kahl VFS, Sobinoff AP, Ling VY, Jebaraj BMC, Janardhanan Y, Haldar R, Bray LJ, Bullinger L, Heidel FH, Kennedy GA, Hill MM, Pickett HA, Abdel-Wahab O, Hartel G, Lane SW
Nat Cancer 2024, 5(1), 47-65 - Dietary restriction interventions: lifespan benefits need resilience and are limited by immune compromise and genetics.
Chen Y, Malik A, Rudolph KL
Signal Transduct Target Ther 2024, 9(1), 335 - Dominant immune tolerance in the intestinal tract imposed by RelB-dependent migratory dendritic cells regulates protective type 2 immunity.
Geiselhöringer AL, Kolland D, Patt AJ, Hammann L, Köhler A, Kreft L, Wichmann N, Hils M, Ruedl C, Riemann M, Biedermann T, Anz D, Diefenbach A, Voehringer D, Schmidt-Weber CB, Straub T, Pasztoi M, Ohnmacht C
Nat Commun 2024, 15(1), 9143 - Denervation alters the secretome of myofibers and thereby affects muscle stem cell lineage progression and functionality.
Henze H, Hüttner SS, Koch P, Schüler SC, Groth M, von Eyss B, von Maltzahn J
NPJ Regen Med 2024, 9(1), 10