Teilbereich 1: Altern von Stammzellen
Die Forschungsgruppen innerhalb des Teilbereichs 1 untersuchen die Ursachen und Folgen der Stammzellalterung. Die Forschungsarbeit erstreckt sich von Modellorganismen über genetische Mausmodelle bis hin zu Mausmodellen, die mit menschlichen Stammzellen angereichert sind.
Mit der Schließung von zwei Gruppen in 2016 ist die Stammzellforschung an Wirbellosen-Modellen im Teilbereich 1 reduziert. Das Institut geht davon aus, dass mit der Rekrutierung neuer Gruppen diese Lücke geschlossen warden kann.
Die Forschung ist durch vier Schwerpunkte definiert:
- Zellintrinsische Mechanismen, die die Funktion alternder Stamm- und Vorläuferzellen einschränken,
- Alternsbedingte Veränderungen der Stammzellnischen und des systemischen Umfelds,
- Mechanismen der klonalen Selektion und epigenetische Drifts bei der Stammzellalterung und
- Mikrobiota- und stoffwechselbedingte Beeinträchtigungen der Stammzellfunktion während des Alterns (im Zusammenhang mit dem neuen Schwerpunkt Mikrobiota und Altern, der ím Teilbereich 2 aufgebaut wird).
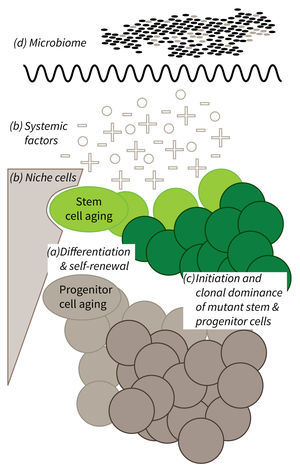
Forschungsfokus von Teilbereich 1.
a) Derzeit ist noch nicht vollständig bekannt, welche Mechanismen die Zellfunktionen im Alter beeinträchtigen. b) Der relative Einfluss von Nischenzellen und systemisch wirkenden Faktoren auf die Stammzellalterung müssen für verschiedene Gewebe noch erforscht werden. c) Die klonale Ausbreitung mutanter Zellen wird mit der Entstehung von Krankheiten im Alter in Verbindung gebracht. Mechanistisch ist der Prozess jedoch noch kaum verstanden. Die Veränderungen in der Farbintensität stehen für die klonale Dominanz von ursprünglichen Stamm- (grün) und Vorläuferzellen (grau). d) Es gibt zunehmend Hinweise darauf, dass alternsbedingte Veränderungen im Mikrobiom die Stammzellfunktion beeinflussen und vice versa.
Publikationen
(seit 2016)
2023
- Human gene-engineered calreticulin mutant stem cells recapitulate MPN hallmarks and identify targetable vulnerabilities.
Foßelteder J, Pabst G, Sconocchia T, Schlacher A, Auinger L, Kashofer K, Beham-Schmid C, Trajanoski S, Waskow C, Schöll W, Sill H, Zebisch A, Wölfler A, Thomas D, Reinisch A
Leukemia 2023, 37(4), 843-53 - Lentiviral in situ targeting of stem cells in unperturbed intestinal epithelium.
Garside GB, Sandoval M, Beronja** S, Rudolph** KL
BMC Biol 2023, 21(1), 6 ** co-corresponding authors - Molecular-defined clonal evolution in patients with classical myeloproliferative neoplasms.
Hinze A, Rinke J, Crodel CC, Möbius S, Schäfer V, Heidel FH, Hochhaus A, Ernst T
Br J Haematol 2023, 202(2), 308-17 - A dysfunctional miR-1-TRPS1-MYOG axis drives ERMS by suppressing terminal myogenic differentiation.
Hüttner SS, Henze H, Elster D, Koch P, Anderer U, von Eyss B, von Maltzahn J
Mol Ther 2023, 31(9), 2612-32 - Author Correction: IFNγ-Stat1 axis drives aging-associated loss of intestinal tissue homeostasis and regeneration.
Omrani* O, Krepelova* A, Rasa SMM, Sirvinskas D, Lu J, Annunziata F, Garside G, Bajwa S, Reinhardt S, Adam L, Käppel S, Ducano N, Donna D, Ori A, Oliviero S, Rudolph KL, Neri F
Nat Commun 2023, 14(1), 6302 * equal contribution - IFNγ-Stat1 axis drives aging-associated loss of intestinal tissue homeostasis and regeneration.
Omrani* O, Krepelova* A, Rasa SMM, Sirvinskas D, Lu J, Annunziata F, Garside G, Bajwa S, Reinhardt S, Adam L, Käppel S, Ducano N, Donna D, Ori A, Oliviero S, Rudolph KL, Neri F
Nat Commun 2023, 14(1), 6109 * equal contribution - Gene regulatory network inference with popInfer reveals dynamic regulation of hematopoietic stem cell quiescence upon diet restriction and aging.
Rommelfanger* MK, Behrends* M, Chen Y, Martinez J, Bens M, Xiong L, Rudolph** KL, MacLean** AL
bioRxiv 2023, 10.1101/2023.04.18.537360 * equal contribution, ** co-corresponding authors - Dynamic DNA methylation reveals novel cis-regulatory elements in mouse hematopoiesis.
Schönung M, Hartmann M, Krämer S, Stäble S, Hakobyan M, Kleinert E, Aurich T, Cobanoglu D, Heidel FH, Fröhling S, Milsom MD, Schlesner M, Lutsik P, Lipka DB
Exp Hematol 2023, 117, 24-42.e7 - Cre recombinase expression cooperates with homozygous FLT3 internal tandem duplication knockin mouse model to induce acute myeloid leukemia.
Straube* J, Eifert* T, Vu T, Janardhanan Y, Haldar R, von Eyss B, Cooper L, Bruedigam C, Ling VY, Cooper E, Patch AM, Bullinger L, Schnoeder TM, Bywater M, Heidel* FH, Lane* SW
Leukemia 2023, 37(4), 741 * equal contribution - Immunoproteasome function maintains oncogenic gene expression in KMT2A-complex driven leukemia.
Tubío-Santamaría N, Jayavelu AK, Schnoeder TM, Eifert T, Hsu CJ, Perner F, Zhang Q, Wenge DV, Hansen FM, Kirkpatrick JM, Jyotsana N, Lane SW, von Eyss B, Deshpande AJ, Kühn MWM, Schwaller J, Cammann C, Seifert U, Ebstein F, Krüger E, Hochhaus A, Heuser M, Ori A, Mann M, Armstrong SA, Heidel FH
Mol Cancer 2023, 22(1), 196