Jena/Los Angeles. Als Abfolge bestimmter Basenpaarungen (Sequenz) ist in der DNA unsere Erbinformation gespeichert. Diese wird weitgehend unverändert von einer Generation an die nächste weitergegeben. Doch wie kann es sein, dass sich die Zellen eines Menschen in Form und Funktion so massiv unterscheiden, wie beispielsweise im Blut, den Nerven, der Haut oder im Zahn, obwohl sie doch alle das gleiche Erbgut (Genom) besitzen? Hier kommt die so genannte „Epigenetik“ ins Spiel. Damit sind vor allem chemische Modifikationen unseres Genoms gemeint, die letztlich beeinflussen, ob und wie eine Zelle auf ihr Erbgut zurückgreifen kann. Das heißt, epigenetische Mechanismen legen fest, wann welches Gen abgelesen oder stummgeschalten wird. Zu den wichtigsten epigenetischen Modifikationen gehören die DNA-Methylierung und die Modifikation von spezialisierten, genom-assoziierten Proteinen (Histonen). Derartige Veränderungen werden auch als Epigenom bezeichnet. Im Gegensatz zum Genom, kann sich das Epigenom rasch verändern und damit auf Veränderungen (z.B. Umwelteinflüsse) reagieren. Die Untersuchung des Epigenoms hat daher die Mechanismen im Fokus, die die Aktivität von Genen steuern, ohne die Basensequenz der DNA zu ändern.
Obwohl die Erforschung des Epigenoms mehr und mehr an Bedeutung gewinnt, ist bisher wenig darüber bekannt, wie sich epigenetische Modifikationen über längere Zeiträume hinweg verändern. Auch fehlt es bisher an systematischen Untersuchungsstrategien. In Kooperation mit dem Humangenetiker Prof. Steve Horvath der University of California, Los Angeles, USA, haben Forscher des Jenaer Leibniz-Instituts für Alternsforschung – Fritz-Lipmann-Institut (FLI) aus der Forschungsgruppe „Bioinformatik für Alterungsprozesse“ und der Core Facility Life Science Computing nachgewiesen, dass sich klassische phylogenetische Methoden systematisch auch auf epigenetische Daten übertragen lassen.
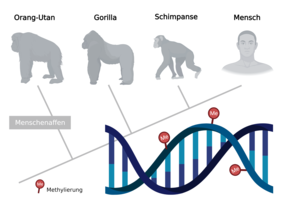
Phylogenetische Stammbäume auf der Basis epigenetischer Daten
In der Biologie werden seit mehr als 50 Jahren auf der Grundlage von sequenzbasierten Methoden phylogenetische Stammbäume zur Rekonstruktion der Evolutionsgeschichte und umfassenden Beschreibung von Verwandtschaftsverhältnissen erstellt. „Bei der Verwendung von epigenetischen Daten fragten wir uns, ob zum einen die darin enthaltenen Informationen eine aussagekräftige evolutionäre Analyse gestatten und zum anderen, ob die Signale über eine längere evolutionäre Distanz hinweg überhaupt gut genug konserviert sind, um aus ihnen phylogenetische Beziehungen richtig rekonstruieren zu können“, erläutert Dr. Arne Sahm den Forschungsansatz.
„Um einen Einblick in die Funktion einzelner Gene und ihrer Stoffwechselpfade zu bekommen, verwendeten wir in der Vergangenheit verschiedene sequenzbasierte Methoden, die wir nun systematisch auf die Ebene der DNA-Methylierung übertrugen“, ergänzt Dr. Sahm, Erstautor der in „Molecular Biology and Evolution“ veröffentlichten Studie. Die DNA-Methylierung ist eine direkte chemische Veränderung von DNA-Basen, die in einem komplizierten Prozess von spezialisierten Enzymen vorgenommen wird. Besonders folgenreich sind Veränderungen im Startbereich von Genen: durch die Methylierung kann die Aktivität eines Gens direkt gesteuert werden.
Epigenetik ermöglicht zuverlässigere Rekonstruktion von Abstammungsverhältnissen
Da sich unsere nächsten Verwandten - die Schimpansen, Gorillas und Orang-Utans - in Bezug auf ihr Genom nur geringfügig vom Menschen unterscheiden, nutzten die Forscher für ihre Analysen einen Datensatz von Blutproben von Menschenaffen. „Wir konnten zeigen, dass die Rekonstruktion von phylogenetischen Bäumen auf der Basis von DNA-Methylierungsdaten sehr gut funktioniert,“ berichtet Prof. Steve Hoffmann, Forschungsgruppenleiter am FLI und Professor für Computational Biology an der Friedrich-Schiller-Universität Jena. „Wir waren besonders überrascht, dass sich die Abstammungsverhältnisse der Menschenaffen mit den epigenomischen Daten noch zuverlässiger rekonstruieren lassen, als mit den genomischen. Da die Methylierung durch Umweltfaktoren oder Krankheiten beeinflusst wird, war das so nicht zu erwarten.“
DNA-Methylierung stark konserviert
Die Forscher vom FLI konnten darüber hinaus nachweisen, dass sich die DNA-Methylierung, zwischen den untersuchten Menschenaffen-Arten nur sehr geringfügig unterscheidet. So sind Genomabschnitte, die im Menschen stark methyliert sind, typischerweise auch in den Menschenaffen durch die Modifikation verändert. Das Gleiche gilt für Teile des Erbguts, die wenig oder gar nicht methyliert sind. Offenbar sind diese epigenomischen Muster über Millionen von Jahren hinweg in der Evolution weitgehend unverändert bestehen geblieben, d.h. stark konserviert. „Die Auflösung der Konservierung ist dabei so hoch, dass sich zwei äquivalente DNA-Bausteine verschiedener Menschenaffenarten hinsichtlich ihrer Methylierung im Mittel stärker ähneln, als zwei direkt benachbarte DNA-Bausteine in der gleichen Art", unterstreicht Prof. Hoffmann.
Einblicke in gewebespezifische phylogenetische Informationen
Anders als das Genom, das in allen Zellen eines Individuums nahezu identisch ist, kann das Epigenom gewebespezifische phylogenetische Informationen widerspiegeln. Daher bieten Vergleiche zwischen verschiedenen Spezies die Möglichkeit, Einblicke in die Evolution von Geweben und nicht nur des gesamten Organismus zu gewinnen. Die Einbeziehung anderer epigenetischer Daten, wie z.B. der Modifikationen von Histonen, könnten hilfreich sein, um derartige Prozesse zukünftig besser zu beleuchten.
Potential für tiefere Einblicke in evolutionäre Unterschiede und Gemeinsamkeiten
„Die Anwendung und Weiterentwicklung der von uns entwickelten Methoden verspricht neue Einblicke in die Mechanismen der epigenetischen Genregulation und in die Entstehung neuer Phänotypen, die auf diesen Mechanismen beruhen,“ erklärt Dr. Sahm. Die Methoden könnten dabei helfen, solche Teile des Erbguts zu identifizieren, die mit großer Wahrscheinlichkeit eine wichtige Rolle bei der Genregulation spielen, weil sie sich über Millionen von Jahren kaum verändert haben. „Umgekehrt können wir mit der beschriebenen Methode auch nach Abschnitten des Epigenoms suchen, die sich in bestimmten Arten im Verlauf der Evolution besonders schnell verändert haben, um zu untersuchen, wie diese epigenetischen Veränderungen das Hervorbringen von Phänotypen beeinflussen“.
Publikation
An analysis of methylome evolution in primates. Sahm A, Koch P, Horvath S, Hoffmann S. Mol Biol Evol. 2021, 38(11), 4700-4714. doi: 10.1093/molbev/msab189. https://academic.oup.com/mbe/article/38/11/4700/6310173
Kontakt
Dr. Kerstin Wagner
Presse- und Öffentlichkeitsarbeit
Tel.: 03641-656378, E-Mail: presse@~@leibniz-fli.de