Das Team der Core Facility Life Science Computing des Leibniz-Institut für Alternsforschung - Fritz-Lipmann-Institut e.V. (FLI) feiert die Veröffentlichung seines neuen Tools, IntersectMe.
Mit diesem leistungsstarken, interaktiven Web-Tool ist der Vergleich und die Analyse mehrerer Datensätze so einfach wie nie zuvor. Ganz gleich, ob man als Biologe überlappende Gene in verschiedenen Experimenten untersuchen oder als Sozialwissenschaftler Interaktionsnetzwerke erforschen möchte.
Das Tool wurde von Dr. Bilal Mustafa, Postdoc in der Core Facility Life Science Computing, entwickelt. In der Core Facility selbst, geleitet von Dr. Karol Szafranski, entstand die Idee aus der Suche nach einem einfachen Tool für die Visualisierung von Genanalysen.
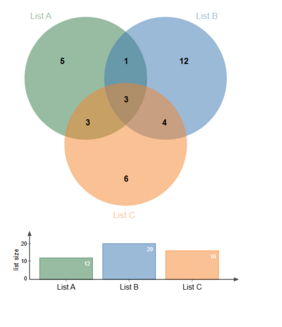
IntersectMe bietet zwei Grundtypen der grafischen Darstellung von Überlappungen: entweder als Venn-Diagramm – für maximal fünf Listen – oder alternativ als UpSet-Plot. Die erzeugten Diagramme lassen sich direkt als publikationsreife Abbildungen in den Formaten PNG, SVG oder als CSV-Liste herunterladen.
Ein weiteres Angebot von IntersectMe ist die Anwendung von Mengenfunktionen, um neue Sublisten zu erzeugen. Möglich ist die Erzeugung von Schnittmengen (engl. intersections), Vereinigung, exklusiven Mengen (symmetrische Differenz) oder orientierter Differenz.
Generell wird das Datenschutzinteresse der Nutzer respektiert. Denn alle Daten, die an den IntersectMe-Server geschickt werden, werden nach Verarbeitung sofort an den Client-Rechner zurückgesendet und vom Server gelöscht.
Die Bedienung von IntersectMe ist intuitiv und benutzerfreundlich. Zahlreiche Parameter sind einstellbar, um Grafiken nach gewünschten Präferenzen darstellen zu lassen. Für die Benutzung ist keine Registrierung nötig.
Testen Sie das neue Tool, welches Sie unter folgendem Link finden: