Forschungsgruppe Hoffmann
Zusätzliche Projekte
Verbundprojekt „de.NBI – Partner: de.STAIR“
Bioinformatics Services for Structured Analysis and Integration of RNA-Seq experiments (de.STAIR)
Der Forschungsschwerpunkt von de.STAIR liegt auf RNA-Sequenzierungs-Analysen mit dem Ziel, Best-Practices und nutzerfreundliche Workflows für die Verarbeitung und Integration von Genexpressionsdaten zu entwickeln sowie Hilfestellung bei der Vorbereitung von Sequenzierexperimenten, der anschließenden Datenanalyse und Interpretation zu geben. Die große Heterogenität in RNA-Seq-Studiendesigns und untersuchten Organismen hat großen Einfluss auf die nachfolgende bioinformatische Analyse. Als Teil des RNA-Bioinformatik-Zentrums (RBC), das die Forschung mit RNA-Bezug innerhalb de.NBI unterstützt, vereint de.STAIR die Expertise von drei beteiligten Arbeitsgruppen, um eine flexible RNA-Seq-Analysis-Workbench für verschiedene RNA-Seq-Protokolle zu entwickeln und zu pflegen, die lokal oder in einer Cloudumgebung (wie z.B. der Open-Source-Plattform Galaxy) genutzt werden kann. Darüber hinaus bietet de.STAIR Workshops, Weiterbildungen und Screencasts für Experten und Anfänger an, um den Zugang zu RNA-Seq-Analysen zu erleichtern.
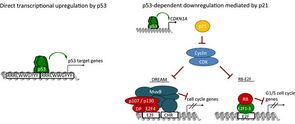
Abbildung des indirekten p53-Genregulierungsnetzwerks
Der Forschungsschwerpunkt des Projektes liegt darauf, Genregulationsnetzwerke zu analysieren, die Zellzyklus und Funktionen des Tumorrepressors p53 beeinflussen. Der Transkriptionsfaktor p53 dient als zentraler Repressor der Tumorentwicklung. Er kontrolliert Zellproliferation und Apoptose durch die Steuerung einer Vielzahl an Zielgenen. Allerdings ist unklar, wie p53 viele dieser Zielgene direkt reguliert, welche Faktoren neben p53 selbst für eine Hoch- oder Herunterregulierung notwendig sind und wie deren Regulierung normale und Tumorzellen beeinflusst. Unser unvollständiges Bild der molekularen Grundlage p53-abhängiger Genregulation ist bisher eine kritische Lücke in unserem Verständnis der Tumorsuppression.
Kontakt

Steve Hoffmann
Gruppenleiter
+49 3641 656810
steve.hoffmann@~@leibniz-fli.de
Patricia Möckel
Sekretariat
+49 3641 65-6240
patricia.moeckel@~@leibniz-fli.de
Team
| Name | Telefon | Tätigkeit | |
|---|---|---|---|
| Steve Hoffmann | +49 3641 656810 | Gruppenleiter | |
| Martin Fischer | +49 3641 656876 | Wissenschaftlicher Mitarbeiter | |
| Konstantin Riege | +49 3641 656875 | Wissenschaftlicher Mitarbeiter | |
| Alena van Bömmel | +49 3641 656868 | Wissenschaftliche Mitarbeiterin | |
| Maja Kinga Olecka | +49 3641 656877 | Postdoc | |
| Kanstantsin Siniuk | +49 3641 656875/6243 | Postdoc | |
| Atakan Ayden | +49 3641 656874 | Doktorand | |
| Aleksandr Cherkasov | +49 3641 656876 | Doktorand | |
| Setenay Gupse Özcan | --- | Doktorandin | |
| Tushar Patel | +49 3641 656868 | Doktorand | |
| Katjana Schwab | +49 3641 656877 | Doktorandin | |
| Robert Schwarz | +49 3641 656057 | Wissenschaftler | |
| Niels Jahn | +49 3641 656049 | Forschungsingenieur | |
| Silke Förste | +49 3641 656243 | Technische Assistentin | |
| Tycho Kirchner | --- | Masterstudent | |
| Jonas Mewes | --- | Bachelorstudent |
* unvollständig aufgrund DSGVO