Forschungsgruppe Hoffmann
Bioinformatik für Alterungsprozesse:
Der Algorithmus des Alterns
Die Forschungsgruppe “Bioinformatik für Alternsforschung” ist eine der jüngsten Gruppen am FLI. Unser Hauptanliegen ist es, zu einem besseren Verständnis beizutragen, wie das Epigenom Prozesse der Genexpression und Maturierung steuert. Dafür entwickeln wir eigene Methoden zur Analyse großer, multidimensionaler biologischer Datensätze. Für unsere Arbeit kooperieren wir mit vielen Forschungsgruppen und sind Mitglied verschiedenster hochkarätiger internationaler Konsortien wie dem International Cancer Genome Consortium (ICGC) oder den BLUEPRINT-Konsortium.
Nachfolgend geben wir einen Einblick in unsere Forschungsschwerpunkte.
Forschungsschwerpunkte
Die Ergänzung epigenetischer Informationen, wie DNA-Methylierung und Histonmodifikationen, um Transkriptomdaten liefert neue Erkenntnisse in epigenetischen Kontrollmechanismen.
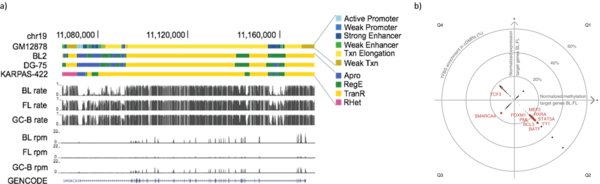
Als Mitglied der deutschen Konsortien ICGC-MMMLSeq und ICGC-DE sowie HNPCCSys und BLUEPRINT helfen wir dabei, hunderte Datensätze unterschiedlicher Tumortypen zu analysieren. Ziel ist es, gemeinsame epigenetische Mechanismen in verschiedenen Geweben, wie bspw. differenziert methylierte Regionen (DMRs), zu identifizieren.
DNA-Methylierung ist eine der häufigsten epigenetischen Veränderungen. Unsere Alignment Software für Reads aus Sequenzierexperimenten (Hoffmann et al., 2009) wurde dahingehend erweitert auch Bisulfit-Sequenzierungsdaten zu mappen (Otto et al., 2013). Dies ermöglicht das Schlussfolgern auf den Methylierungsgrad anhand von gemappten Reads.
Um signifikante Unterschiede des Methylierungsgrades zwischen verschiedenen Bedingungen und in einer großen Anzahl von Proben zu finden, haben wir die Software metilene (Juehling et al., 2016) entwickelt, die in der Lage ist, sehr schnell und mit hoher Zuverlässigkeit differentiell methylierte Regionen (DMRs) zu identifizieren.
Bei der RNA-Sequenzierung stammen die Sequenzstücke aus mRNAs oder ncRNAs und können mehrere Exons abdecken, d.h. Exon-Übergänge überspannen. Der biologische Prozess, der zur Entstehung von Exon-Verknüpfungen führt, ist als Splicing bekannt. Unsere Software Segemehl ist in der Lage, mit solchen Splicing-Vorgängen umzugehen, sie zu detektieren und auszugeben. Um dabei Fälle von alternativem Splicing aufzuspüren, die oft eine wichtige Rolle bei der Differenzierung oder der Entstehung von Krankheiten spielen, haben wir die Software DIEGO (Doose et al., 2017) entwickelt. Die Ergebnisse von DIEGO waren schon oft essentielle Grundlage für eine Reihe unserer Publikationen zu Lymphomdatensätzen.
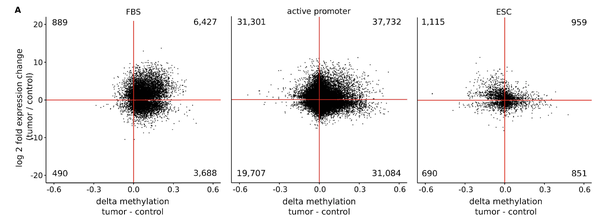
In einem unserer jüngsten Projekte haben wir den bivalenten Zustand von Chromatin in Krebszellen untersucht. Bivalentes (balanciertes oder pausierendes) Chromatin vereint transkriptionsaktivierende und -unterdrückende Histonmodifikationen in einer spezifischen Region, wie einem Promoter oder Enhancer. Eine spezielle Kombination solch epigenetischer Marker balanciert die Transkriptrate auf einem niedrigen Level und hält somit die Gene für eine schnelle Expression bereit. Typischerweise ist die DNA an bivalenten Promotoren in gesunden Zellen nur gering methyliert, in Krebszellen ist der Methylierungsgrad hingegen erhöht. Wir haben aus Chromatindaten einen Klassifikator entwickelt, mit dem Krebs allein über die Hypermethylierung des bivalenten Chromatins identifiziert werden kann. Getestet an mehr als 7.000 DNA-Methylierungs-Datensätzen unterschiedlicher Krebsarten, erreicht unsere Vorhersage eine Zuverlässigkeit von 0,92 AUC (Fläche unter der ROC-Kurve). Obwohl eine verstärkte DNA-Methylierung oft mit transkriptionellem Gen-Silencing verbunden wird, konnten kürzlich auch scheinbar widersprüchliche, statistisch positive Abhängigkeiten zwischen DNA-Methylierung und Epxressionsgraden gezeigt werden. Die kombinierte Re-Analyse von Expressions- und Methylierungsdaten von mehr als 5.000 Proben bestätigte das überraschende Ergebnis, dass die Hypermethylierung von bivalentem Chromatin und die Hoch-Regulierung des entsprechenden Gens ein generelles Phänomen in Krebzellen ist. Derartige Prozesse steuern viele verschiedene Entwicklungsgene und Transkriptionsfaktoren inklusive dutzender Homeobox-Gene und andere Gene, die im Umfeld der Transkription von Krebszellen eine Rolle spielen. Wir schlussfolgern daraus, dass die Störung des bivalenten Zustands von Chromatin unmittelbar mit der Entstehung von Tumoren verbunden ist.
Kontakt

Steve Hoffmann
Gruppenleiter
+49 3641 656810
steve.hoffmann@~@leibniz-fli.de
Patricia Möckel
Assistenz
+49 3641 65-6240
patricia.moeckel@~@leibniz-fli.de